Mejorando la predicción del síndrome de Down mediante un modelo de clasificación de datos médicos inteligente- Caso de Estudio
Improving Downs Syndrome Prediction with a Smart Medical Data Classification Model- Case of Study
Juan Jose Saldana-Barrios
ISES-CIDITIC
Facultad de Ingeniería de Sistemas Computacionales
Universidad Tecnológica de Panamá
[email protected]
Tomas Concepción
ISES-CIDITIC
Facultad de Ingeniería de Sistemas Computacionales
Universidad Tecnológica de Panamá
[email protected]
Miguel Vargas-Lombardo
ISES-CIDITIC
Facultad de Ingeniería de Sistemas Computacionales
Universidad Tecnológica de Panamá
[email protected]
Resumen– En el área de la salud la aplicación de medicamentos, realización de cirugías, proyecciones sobre la dispersión de enfermedades infecciosas, estudios del cáncer y otras, características como la precisión y la exactitud son fundamentales. En los últimos años, los métodos de inteligencia artificial conocidos como métodos de aprendizaje de máquinas son cada vez más usados para lograr obtener la mayor precisión y certeza en la predicción y clasificación de datos sensibles para la comunidad médica. Actualmente el método de predicción utilizado para estimar la probabilidad de poseer la Aneuploidía conocida como síndrome de Down utiliza límites inferiores y superiores para indicar si los múltiplos de las medianas conocida como MoMs, son calculados mediante pruebas químicas y se encuentran dentro del rango de una población saludable o anormal. Utilizando estos métodos de aprendizaje de máquinas podemos calcular estos límites dinámicamente. El algoritmo determina los parámetros ajustándose a lo indicado por la misma población mejorando así precisión de la estimación.
En este trabajo primero se propone un modelo para calcular dinámicamente los valores superiores e inferiores que actúan como límite para pronosticar si un paciente presenta o no esta alteración cromosómica. Segundo, el modelo es explicado e implementado y tercero, los resultados obtenidos mediante el método de máquinas de vectores de soporte y clasificadores bayesianos ingenuos son comparados para determinar cuál de los dos proporciona mejores resultados al momento de predecir el riesgo de padecer esta aneuploidía.
Palabras claves– Clasificador Bayesiano Ingenuo, Máquina de Aprendizaje Automático, Máquina de Vectores de Soporte, Síndrome de Down, Salud Electrónica.
Abstract– In health areas like drugs application, surgeries, projection of the spreading of contagious diseases, study of cancer and others, estimation, accuracy and precision are crucial. In the last few years, machine-learning methods have been used to obtain the best precision in prediction and classification of sensitive data for the medical community. Currently the Down’s syndrome risk estimation process uses established inferior and superior limits to determine if a chemical test is normal or abnormal. Using machine-learning methods we can calculate these limits dynamically. It would adapt the process to the parameters of the population improving it´s results.
In this paper we first propose a model to dynamically calculate the values of the upper and lower limits of a healthy population, second the model is implemented and the process is explained and third we compare the results of applying Support Vector Machine and Naive Bayes machine learning methods to predict the risk of having Downs syndrome.
Keywords– Naïve Bayes, Machine Learning, Support Vector Machine, Down’s syndrome, eHealth.
1. Introducción
Hoy en día, la clasificación de información y el aprendizaje de las máquinas son disciplinas científicas altamente estudiadas dentro del campo de la inteligencia artificial, ya que ellas permiten extraer y analizar enormes cantidades de datos lo cual es imposible para un ser humano. Adicionalmente, es común encontrar mucha información útil aún cuando esta no está propiamente clasificada, ni estructurada en bases de datos, como lo son la gran variedad de documentos digitales como exámenes, diagnósticos o recetas. Muchos de los obstáculos encontrados en estas tareas son solucionados utilizando técnicas inteligentes de clasificación de datos. Entre algunas de estas técnicas podemos mencionar las máquinas de vectores de soporte, clasificador bayesiano ingenuo, árboles de decisión, selvas aleatorias y regresión lineal. Dos de estas técnicas la utilizaremos en el contexto del síndrome de Down.
El síndrome de Down, también conocido como trisomía 21, es una aneuploidía causada por la presencia de 3 cromosomas 21. Actualmente, el riesgo de presentar esta enfermedad es calculado utilizando el método de máxima verosimilitud (Likelihood) como base, tomando como punto de inicio la edad de la madre y seguido de algunos factores de corrección como lo son la etnia de la madre, la presencia de diabetes, feto afecto previo entre otros. El segundo método consiste en realizar exámenes químicos analizando la sangre en la madre determinando las MoMs de cada marcador químico. Estos últimos son comparados con un rango de valores superiores e inferiores fijos, estipulados para mujeres latinoamericanas.
En Panamá, especialistas en el campo de cribado prenatal argumentan que una pequeña cantidad de casos de pacientes con síndrome de Down son encontrados en la población indígena de nuestro país. Esto los conlleva a suponer que la variable etnia posee un gran impacto en la estimación del riesgo de poseer la enfermedad y muy probablemente los resultados emitidos por las actuales pruebas deban ser sometidos a un factor de corrección cuando el paciente presenta la etnia indígena, factor que para la etnia indígena del país no se ha calculado aún.
En trabajos anteriores [1], un modelo para la predicción del síndrome de Down fue propuesto. Uno de los componentes del modelo, el cual se encarga de realizar el cálculo del riesgo ha sido implementado y validado sin la utilización de límites superior e inferior fijos. Nuestra intención es poder comparar los resultados emitidos por dicho componente para estimar la presencia de esta aneuploidía utilizando los parámetros poblacionales de mujeres embarazadas de la localidad, aplicando métodos de inteligencia artificial.
El resto del documento está estructurado de la siguiente manera: Sección 2 presenta un estudio del arte sobre la trisomía 21. Sección 3 y 4 explican el funcionamiento de las técnicas conocidas como máquinas de vectores de soporte y clasificadores bayesianos ingenuos como métodos de clasificación de datos. Sección 5 explica como estos métodos son implementados en el modelo propuesto. La sección 6 presenta los resultados de las pruebas y finalmente la sección 7 las conclusiones y trabajos futuros.
2. Antecedentes
Tal y como se menciona en [1], [2], la trisomía 21, también conocida como síndrome de Down, es una aneuploidía o desorden cromosómico donde el feto presenta 3 cromosomas 21. Esta enfermedad es responsable de múltiples discapacidades físicas en el paciente, algunas de las cuales conllevan hasta la muerte del bebé antes de su nacimiento. Las madres cuyos bebes padecen de esta enfermedad presentan problemas prenatales. El síndrome de Down causa defectos cardíacos, características faciales específicas, bajo tono muscular, malformaciones de algunos órganos, crecimiento retardado, déficit mental, desórdenes de audición, visión y otras enfermedades como el Alzheimer.
2.1 Trisomía 21
El tamizaje prenatal, es una prueba sanguínea realizada a la madre del feto para determinar si el mismo presenta el riesgo de padecer de síndrome de Down, defecto de tubo neural, epidermólisis Bullosa, trisomía 18, espina bífida y preeclampsia. El tamizaje o cribado no es un diagnóstico como tal, pero permite predecir la presencia de alguna de estas enfermedades. Si los resultados muestran un algo riesgo de padecer alguna de estas anomalías, se procede a realizar una prueba de tipo invasiva en el feto conocida como amniocentesis, un procedimiento de diagnóstico que extrae líquido amniótico procedente del saco amniótico del útero de la madre insertando una aguja siendo este algo riesgoso para el feto. Mientras más exacta es la prueba, menor es la cantidad de pruebas con casos falsos positivos y falsos negativos disminuyendo así la necesidad de realizar amniocentesis.
Esta prueba es realizada actualmente en el primer y segundo trimestre de gestación siendo más efectiva y más común su realización en el segundo trimestre debido a los cambios químicos en la sangre materna son más pronunciados después de la semana 11. Los marcadores químicos a medir son los siguientes:
Alfa-fetroproteína (AFP) es una proteína producida por el feto en el hígado y en el saco amniótico. Esta proteína se incrementa hasta la semana 12 de gestación para luego disminuir hasta que el bebé nace. Como la madre y el feto están ligados a través de la placenta, esta proteína puede cruzar a la madre y aparecer en su sangre. Una elevada cantidad de AFP en la sangre materna puede indicar que el feto padece de un problema médico. Pruebas para medir los niveles de la AFP son realizados para determinar la presencia de trisomía 18 o 21 como parte del triple test o para detectar defectos de tubo neural.
La Gonadotropina coriónica humana (hCG) es una hormona producida durante el embarazo por el embrión inmediatamente luego de la implantación. Muchos de los test miden la presencia de β como subunidad de hCG, de manera que los niveles no se confunden con otras hormonas similares como lo son la hormona luteinizante y la luteoestimulante. En las pruebas sanguíneas, la βhCG puede ser detectada y casi siempre indican embarazo. Una baja presencia de esta hormona puede indicar existencia de trisomía 18 mientras que una alta concentración de la misma, la presencia de trisomía 21. Estriol unconjugado (uE3) es un estrógeno encontrado principalmente en la placenta durante el embarazo. Esta puede ser medida cuando la hormona pasa de la placenta a la sangre de la madre. Cuando los niveles de esta hormona son muy bajos, indican la presencia de trisomía 21 y 18.
Tabla 1. Patrón de análisis de los indicadores químicos de un cribado prenatal del segundo trimestre de gestación
Riesgo para: |
AFP |
UE3 |
hCG |
| Síndrome de Down |
Bajo |
Bajo |
Elevado |
| Síndrome de Edward |
Bajo |
Bajo |
Bajo |
| Espina Bífida |
Elevado |
Normal |
Normal |
| Anencefalia |
Elevado |
Bajo |
Normal |
Luego que el cribado es realizado los resultados son comparados con los múltiplos de las medianas establecidos para una región específica. Cuando estos valores exceden los límites y las combinaciones de estos marcadores concuerdan con la tabla anterior, el test se considera positivo.
Para que una prueba sea considerada significativa, esta debe de alcanzar una tasa de detección (detection rate DR) mayor al 75% y 3 de falsos positivos, porcentajes establecidos por el Comité Nacional de Cribado Prenatal en el 2008. Para que el cribado de trisomía 21 sea considerado preciso, variable como el peso de la madre, su etnia y la presencia de diabetes y hábitos como el fumar deben ser tomados en cuenta ya que estos afectan la exactitud del test. La siguiente ecuación muestra el calculo de la MoM para cada marcador:
Por ejemplo: Si un paciente tiene un βhCG de 80,456 y la media de la población en esa semana de gestación es de 28,734 la MoM del paciente es 2.8, más del doble de la media de la población. Por ende, al paciente se le debe hacer un test físico para determinar la presencia de trisomía 21.
2.2 Clasificador Bayesiano Ingenuo
El clasificador bayesiano ingenuo(NB) es un clasificador probabilístico utilizado en el aprendizaje de máquinas el cual utiliza el teorema de Bayes pero con la característica de asumir independencia entre las variables involucradas. Es decir, las variables no tienen dependencia una con la otra al momento de realizar la clasificación de los datos. NB necesita para su parametrización, un grupo de data previamente clasificada, al cual se le denominará grupo de entrenamiento. Este conjunto de datos es analizado y procesado para la creación de un algoritmo llamado modelo. Este modelo posteriormente, es el que recibirá data no clasificada para clasificarla en base a las reglas del modelo clasificador. En la mayoría de los casos, las reglas son una variante del teorema de Bayes en el cual cada variable es mutuamente independiente de la otra, simplificando el proceso al momento de calcular las probabilidades de cada clase.
Entre algunas de las ventajas de utilizar NB podemos mencionar que este método es relativamente fácil de implementar y provee una buena exactitud en la mayoría de los casos incluso cuando el conjunto de datos de entrenamiento utilizado sea pequeño.
Entre algunas de las desventajas podemos mencionar el hecho de que si existe dependencia entre las características analizadas, esto puede afectar la clasificación de los datos. Es importante verificar el conjunto de datos si existe dependencia.
2.3 Máquinas de vectores de soporte
Las máquinas de vectores de soporte (Support Vector Machine-SVM) [3] son una familia de máquinas de aprendizaje como los clasificadores bayesianos ingenuos. Estos necesitan un conjunto de datos de entrenamiento para poder generar un modelo que permita clasificar los datos en una u otra categoría. A diferencia de NB, las máquinas de vectores de soporte son clasificadores no probabilísticos ya que estos clasifican en base a posición de la data en un plano vectorial, mas no así con modelos de grupos probabilísticos.
do un conjunto i de características, el modelo funciona en i dimensiones de espacio vectorial. Un vector, es este contexto, puede ser considerado un dato que posee i tipos de valores más el valor de la clase, como en una lista d=<V1,V2,V3,. . . ,Vi,Vc>. sadoen un conjunto de datos, SVM calcula los hiperplanos, siendo estos un número menor en cantidad que el número de características a evaluar, para poder clasificar las clases. Estos hiperplanos deben alcanzar la mayor cobertura posible para ser precisos al momento de separar los datos. En los márgenes de estos hiperplanos residen los vectores de soporte, los cuales se encuentran en los bordes de los grupos de clases. En la figura 1 se puede apreciar un ejemplo de un hiperplano. Usualmente, los hiperplanos son calculados con márgenes poco sensibles [3] donde algunos datos no son tomados en consideración para poder dibujar un hiperplano en base a la mediana de los grupos. Esto se da más que todo cuando los datos son linealmente separables. Sin embargo, cuando no lo son, SVM usa la funciones Kernel para transformar el vector de espacio para hacerlo más linealmente separable y calcular los hiperplanos.
La inteligencia artificial está siendo fuertemente utilizada para la clasificación de registros médicos. Por ejemplo [4] se utilizan las redes neuronales artificiales para estimar el riesgo de una aneuploidía trabajando con 9 variables y 51,208 ejemplos de entrenamiento y 16,898 muestreos de prueba. Otros casos como en [5] la información del Kinect es utilizada con SVM para detectar cuándo es más probable que una persona de tercera edad tenga una caída midiendo sus pasos y analizado los cambios en su postura de estar sentado a estar de pie y viceversa. En [6] el clasificador predice la aparición de cáncer de pulmón con un 95.7% de exactitud; en [7] y [8] se obtuvo un 69% y 97.37% de exactitud respectivamente al medir el concepto de la calidad de vida de los pacientes utilizando cuestionarios para conocer sus opiniones. En [9] los electrocardiogramas son clasificados para detectar ritmos cardiacos anormales con una precisión del 98.4%; se [10] procede a clasificar la comida utilizando cámaras para automatizar el cálculo del consumo alimenticio diario del paciente con un 92.23% de exactitud en base a color, textura, tamaño y forma de la comida; en [11] la incidencia de malaria en Mozambique es calculada con una desviación estándar de error 0.0032669 en las pruebas; se [12] implementa un modelo de predicción para ayudar a identificar grupos de pacientes (HRQoL) que requieren intervención, con un porcentaje de exactitud de 93%; en [13] un modelo de identificación automática para el estatus de pacientes fumadores de un sistema de registro electrónico no estructurado es desarrollado, con un F1 de 83.66%; en [14] los datos de un sistema de registro electrónico son clasificados para detectar trombo embolismo venoso, alcanzando un área bajo la curva de 98%; [15] propone un modelo de clasificación de abreviaciones ambiguas en notas 94.97% de exactitud; y en otros como en [16], un grupo de SVM con el método de agrupamiento conocido como k-Means Clustering y algoritmos genéticos son utilizados para diagnosticar diabetes en mujeres embarazadas con una exactitud 98.82%. Cada uno de los trabajos aquí mencionados son ejemplos de la utilización de máquinas de aprendizaje automático, utilizadas dentro del contexto de salud.
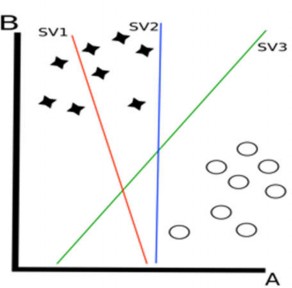
Figura 1. Representación de los hiperplanos (SV1, SV2, SV3) calculados por una máquina de vector de soporte. SV1 no separa las clases, SV2 las separa pero con un pequeño margen, mientras que SV3 los separa con la más amplia brecha posible entre las clases.
Entre algunas de las ventajas que podemos mencionar sobre la clasificación de los datos mediante este método [17] está el hecho de que con los Kernels, SVM nos proporciona flexibilidad para elegir crear un margen más estricto o uno más flexible por medio de parámetros. También que SVM provee una buena generalización para el conjunto de datos de prueba; si los parámetros Costos (C) y los gamma (γ) son definidos correctamente para la funcion radial gausiana, el modelo se mantiene robusto aun cuando el conjunto de datos de prueba tenga un sesgo relativo.
SVM provee una solución única para todo el conjunto de datos y adicional eligiendo el Kernel apropiado, como lo es el Kernel gaussiano, mayor efecto puede aplicarse en las similitudes de los pruebas de ejemplo.
Entre algunas de las desventajas podemos mencionar el hecho de que SVM requiere un mayor poder computacional en comparación a otras técnicas para ejecutar los cálculos, y este requerimiento se incrementa a medida que el conjunto de datos es mayor. La afinación y la elección del Kernel debe ser preciso para lograr obtener una mejor aproximación.
3. Objetivos del proyecto
El principal objetivo de este proyecto es el de proponer un modelo que permita clasificar de una manera dinámica, mediante la utilización de métodos de inteligencia artificial como las máquinas de vectores de soporte y clasificador bayesiano ingenuo, la existencia o no de la trisomía 21, pronosticando la presencia del síndrome de Down dinámicamente, en base a parámetros poblacionales de nuestro país. A continuación, se explica el proceso del análisis desarrollado en esta investigación.
3.1 Desarrollo de los Modelos de Máquinas de Aprendizaje Automático
El modelo está compuesto por dos componentes de estimación, el primer modelo esta basado en el método conocido como clasificador bayesiano ingenuo mientras que el segundo utiliza máquinas de vectores de soporte. Estos modelos tomarán los valores de entrada proveniente de los laboratorios médicos del paciente y pronosticarán la existencia de la enfermedad. Los datos están compuestos de 4 variables: α-Fetoproteina (AFP), Estriol no conjugado(UE3), β-Humana Gonadotropina Coriónicas (BHCG) y la variable a clasificar. La clasificación separa a los individuos en 2 clases:
Normal, en el cual las muestras presentan una condición estable y la anormal, donde los múltiplos de las medianas de los marcadores químicos presentan un rango fuera de lo establecido como normal. La condición anormal, en este caso, hará referencia a la presencia de trisomía 21 o síndrome de Down, ya que este clasificador puede aplicarse a otras enfermedades también estimadas mediante las pruebas de cribados prenatales, como lo son la trisomía 18 o síndrome de Edward, espina bífida y preeclampsia. Estas condiciones son agrupadas dentro de la clasificación anormal.
3.2 Modelo de Detección Bayesiano Ingenuo
Para desarrollar el componente con NB, primero calculamos el promedio de cada característica mediante la ecuación 2:
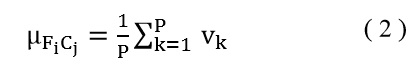
Donde µ FiCj es el promedio de la característica Fi de la clase Cj con una población de p con un valor de V.
Luego se procede a calcular la varianza para cada característica de cada clase aplicando la ecuación 3:
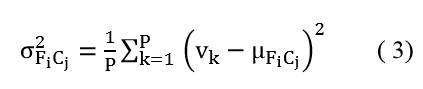
Donde σF2iCj es la varianza característica de Fi de la clase de Cj. La probabilidad de una clase Cj en una población es obtenidamediante la ecuación 4:
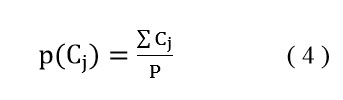
Una vez todas las probabilidades de p( Fi | Ci ) son calculadas, se procederá a calcular la probabilidad de cada funcionalidad:
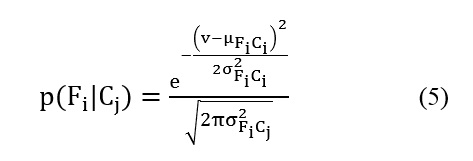
El último paso pudiera ser omitido ya que no es fundamental para hacer la clasificación porque no afecta la probabilidad del resultado. La asunción ingenua aplica:
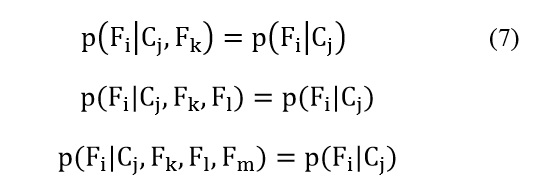
ya que la característica Fi es independiente de las otras características Fk , Fl , Fm , . . . donde i≠k, l , m, . . . , este hace más fácil de calcular las posibilidades de Fi . La probabilidad de la clase en base a dicha característica se calcularía 5:
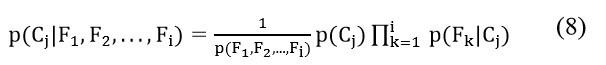
Luego que todas las probabilidades para cada clase son calculadas, el último paso es evaluar que clase tiene la mayor probabilidad de ocurrencia. Esto es realizado mediante la regla de decisión máximo a posteriori.
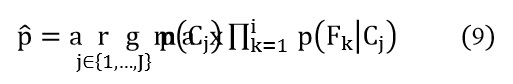
3.3 Modelo de máquina de soporte de estado
La implementación de este componente se llevó a cabo utilizando el lenguaje de programación R. Se utilizó el paquete e1071 [18] el cual es una implementación del paquete libsvm (21) los cuales generan modelos para SVM. Este paquete o librería de R presenta las siguientes características
Clasificación C y v
Regresión v y ε
Métodos de Kernel incluidos: lineal, poli nominal, sigmoidal and funciones de base radial.
El modelo utilizado para la prueba implementa una clasificación C con funciones radiales Gaussianas. Este modelo es fácil de configurar utilizando dos parámetros y provee generalmente un buen desempeño. Los dos parámetros son costo (C) y gamma (γ). El costo es el parámetro para el método de clasificación C el cual le dice al modelo cuan rígido o flexible será el vector de soporte. Cuando el conjunto de datos de entrenamiento presente ruido, el modelo puede omitir algunos datos para poder proveer hiperplanos más anchos que el promedio de las clases, haciendo que el modelo sea más preciso en la clasificación de datos desconocidos.
Tabla 2. Comparación de resultados entre Naive Bayes y Support Vector Machine
|
Naive Bayes |
SVM
| ft: SVM
|
ft: SVM
|
ft: SVM
|
ft: SVM
|
|
| Exactitud |
50.00% |
75.00% |
80.00% |
95.00% |
95.00% |
95.00% |
| Precisión |
33.33% |
75.00% |
100% |
87.50% |
100% |
100% |
| Sensibilidad |
42.86% |
42.86% |
42.86% |
100% |
85.71% |
85.71% |
| MCC |
-0.032 |
0.419 |
0.572 |
0.899 |
0.892 |
0.892 |
Gamma es el parámetro de la función radial gaussiana del kernel el cual puede ser asignado libremente. Éste parámetro determina cuan ancha, o plana, es la distribución de la clase. Mientras más bajo sea el parámetro gama, más plana y angosta se convierte la distribución. Para poder preparar el componente SVM del modelo, el conjunto de datos de entrenamiento y de prueba son cargados en R leyendo un archivo de valores separados por coma mediante la utilización del siguiente comando:

Figura 2. Asignación de datos
La primera línea carga la librería e1071. La segunda y tercera línea cargan los archivos que contienen el conjunto de datos de entrenamiento y de prueba. Una vez esto es realizado, procedemos a crear el modelo con el siguiente comando:

Figura 3. Cargando modelos.
Aquí, V4 es la columna con las variables (la cuarta columna). Los valores de γ y C pueden ser calculados mediante prueba y error donde quiera se ajuste al conjunto de datos de prueba: en todas las pruebas, el valor del costo es 100. Para la predicción, las siguientes instrucciones son necesarias:

Figura 4. Corriendo el modelo.
La variable de predicción almacena el valor que es predecido con el modelo y el conjunto de datos de prueba, donde la cuarta columna es removida para establecer las clases. Luego la variable tab almacena la matriz de confusión de la predicción (asignada como “pred”) y el conjunto de datos de entrenamiento (asignado a “true”).
4. Resultados
Luego que el modelo es creado e implementado, se tomaron 100 muestras médicas seleccionadas al azar por especialistas en cribado prenatal, sin incluir ningún dato que pudiera identificar al paciente de la muestra, para mantener la privacidad de los mismos. Estos datos de prueba fueron insertados en ambos componentes, tanto el de NB como el de SVM.
NB y SVM fueron comparados y analizados tomando en cuanta su exactitud, precisión, sensibilidad y coeficiente de correlación de Matthew. Exactitud es la medida que indica cuan cerca los resultados están del valor esperado. Precisión mide cuan distribuido los resultados están de ellos mismos. Mientras más cerca está el valor estimado del verdadero valor, más alta es la exactitud y más cercanos se encuentran los resultados entre sí, mayor es la precisión. Se espera que los resultados muestren una buena exactitud y precisión para poder obtener un test congruente.
Sensibilidad es la porción de los test verdaderos positivos versus todos los resultados realmente positivos. Se calcula mediante la suma de los test verdaderos positivos y falsos negativos.
El coeficiente de correlación de Matthew (MCC) mide que tan bien se comporta un clasificador en una tarea de clasificación entre variables de dos clases[19] sin importar el tamaño de la muestra o la población. Mientras el índice MCC es más cercano a 1, mejor es la clasificación. Un índice de 0 significa que no existe diferencia entre la predicción realizada mediante el clasificador y una predicción aleatoria. Un índice de -1 indica que la predicción es totalmente opuesta a los resultados reales.
Los resultados fueron calculados con las siguientes ecuaciones:
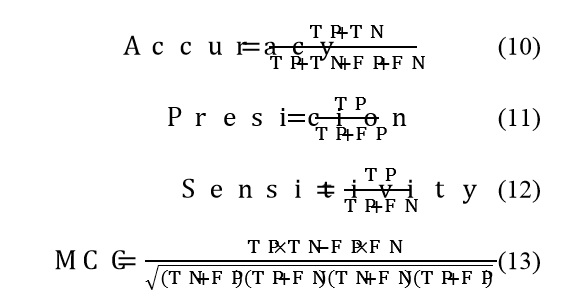
NB obtuvo una precisión menor al realizar la clasificación. SVM obtuvo una exactitud mínima del 75% con un MCC de 0.419 y una exactitud de 95% con MCC de 0.899 respectivamente.
SVM arrojó según lo comparado mejores resultados que NB. Sin embargo, debemos tomar en consideración que el tamaño de la muestra que se utilizó como conjunto de datos de entrenamiento y de prueba tienen influencia sobre el resultado de NB, ya que el mismo utiliza el método de probabilidad entre las clases. A diferencia de NB, SVN genera el modelo con los parámetros establecidos por lo que los datos de entrada son clasificados en base a su posición en el hiperplano donde ellos residen.
5. Discusión
Mediante ambos componentes, se está logrando clasificar el resultado de los pacientes dentro de un rango normal o anormal, de una manera dinámica basándonos en muestreos previamente realizados, sin la necesidad de establecer límites inferiores y superiores estáticos para indicar el rango que determina o no la presencia de una enfermedad en una población.
Los resultados obtenidos pueden ser mejorados, ya que el número de muestras seleccionadas para realizar las pruebas debió ser mayor, pero la disponibilidad de los mismos es una limitante actual. Adicional, el conjunto de datos de entrenamiento fue seleccionado del muestreo y probablemente no es el mejor. Para poder obtener resultados más precisos, debemos contar con el apoyo de instituciones y especialistas que nos proporcionen un muestreo mucho mayor y seleccionen más cuidadosamente un conjunto de datos de entrenamiento más sensitivo.
Basado en estos resultados, por el momento podemos argumentar que SVM aplicado en paralelo con los métodos actuales de predicción, puede mejorar la estimación del riesgo de padecer el síndrome de Down propuesta en [1], donde se agrega una variable adicional de geolocalización (variable aún no tomada en cuenta en el proceso de estimación) y un modelo estándar para el compartimiento de la información entre instituciones de salud para obtener un muestreo mayor de datos.
Además, este trabajo continua con datos experimentales proporcionados por otros laboratorios en la provincia de Chiriquí, Panamá. En esta etapa del proyecto, se requiere de mayor cantidad de datos de muestra para poder analizar, validar y certificar con certeza el funcionamiento del modelo. A medida que los componentes se utilicen por los laboratorios, el número de datos de muestras aumentará, por lo que será posible ser cada vez más acertado en la clasificación y predicción basados en los propios parámetros de la población.
6. Conclusiones
Las máquinas de soporte de estado y el clasificador bayesiano ingenuo son métodos de aprendizaje automático de máquinas, los cuales siendo bien implementados ayudan a los especialistas en la salud a tomar mejores decisiones basados en sus experiencias.
La implementación del modelo utilizando NB y SVM mostró resultados aceptables. Pero, sabemos que la calidad de la muestra no es la mejor y el tamaño de la data de muestra es pequeño. Si el mismo modelo es aplicado bajo mejores condiciones, pruebas propuestas para trabajos futuros donde se cuente con mayor cantidad de recursos, los resultados tendrán mayor exactitud y precisión en la predicción.
El contar con un conjunto de datos adecuado es un factor importante independientemente del método de clasificación utilizado, porque los algoritmos dependen de un conjunto de datos de entrenamiento para poder crear el modelo que posteriormente clasificará la data. Este argumento tiene mayor ponderación en algunos métodos de clasificación que en otros.
Mientras se levantaba el estado del arte sobre la utilización de métodos de aprendizaje automático en el campo de la salud y al haberlos aplicados en nuestros modelo, no podemos argumentar que un método es mejor que otro ya que la clasificación dependerá de la naturaleza de las características de los datos a clasificar, de la relación entre las características y el tipo de datos a ser clasificado.
Con este proyecto intentamos mejorar el proceso actual de estimación del síndrome de Down permitiendo así a los médicos proporcionar a sus pacientes un más adecuado y temprano tratamiento para lograr un proceso de parto menos riesgoso y disminuir el uso de métodos invasivos como la amniocentesis los cuales ponen en riesgo al feto.
7. Referencias
[1] J. Saldaña and M. Vargas-Lombardo, “eHealth Management Platform for Screening and Prediction of Down’s Syndrome in the Republic of Panama,” E-Health Telecommun. Syst. Networks, vol. 03, no. 03, pp. 33–42, Sep. 2014.
[2] D. Patterson, “Molecular genetic analysis of Down syndrome,” Hum. Genet., vol. 126, no. 1, pp. 195–214, Jul. 2009.
[3] C. Cortes and V. Vapnik, “Support-vector networks,” Mach. Learn., vol. 20, no. 3, pp. 273–297, 1995.
[4] A. Neocleous, K. Nicolaides, and C. Schizas, “First Trimester Non-invasive Prenatal Diagnosis: A Computational Intelligence Approach,” IEEE J. Biomed. Heal. Informatics, pp. 1–1, 2015.
[5] M. Parajuli, D. Sharma, D. Tran, and W. Ma, “Senior health monitoring using Kinect,” Commun. Electron. (ICCE), 2012 Fourth Int. Conf., pp. 309–312, 2012.
[6] L. G. Ahmad, A. T. Eshlaghy, A. Poorebrahimi, M. Ebrahimi, and A. R. Razavi, “Using Three Machine Learning Techniques for Predicting Breast Cancer Recurrence,” J. Heal. Med. Informatics, vol. 04, no. 02, pp. 1–3, 2013.
[7] C.-K. Chen, M. Bruce, L. Tyler, C. Brown, A. Garrett, S. Goggins, B. Lewis-Polite, M. L. Weriwoh, P. D. Juarez, D. B. Hood, T. Skelton, and D. B. Hood, “Analysis of an Environmental Exposure Health Questionnaire in a Metropolitan Minority Population Utilizing Logistic Regression and Support Vector Machines,” J. Health Care Poor Underserved, vol. 24, no. 1A, pp. 153–171, 2013.
[8] S. Kumar, M. L. Rana, K. Verma, N. Singh, A. K. Sharma, A. K. Maria, G. S. Dhaliwal, H. K. Khaira, and S. Saini, “PrediQt-Cx: post treatment health related quality of life prediction model for cervical cancer patients.,” PLoS One, vol. 9, no. 2, p. e89851, 2014.
[9] R. J. Martis, U. R. Acharya, L. C. Min, K. M. Mandana, a K. Ray, and C. Chakraborty, “Application of higher order cumulant features for cardiac health diagnosis using ECG signals,” Int. J. Neural Syst., vol. 23, no. 4, p. 1350014, 2013.
[10] P. Pouladzadeh, S. Shirmohammadi, and T. Arici, “Intelligent SVM based food intake measurement system,” 2013 IEEE Int. Conf. Comput. Intell. Virtual Environ. Meas. Syst. Appl. CIVEMSA 2013 - Proc., pp. 87–92, 2013.
[11] O. P. Zacarias and H. Bostr, “Comparing Support Vector Regression and Random Forests for Predicting Malaria Incidence in Mozambique,” no. January 2016, pp. 217–221, 2013.
[12] S.-K. Lee, Y.-J. Son, J. Kim, H.-G. Kim, J.-I. Lee, B.-Y. Kang, H.-S. Cho, and S. Lee, “Prediction Model for Health-Related Quality of Life of Elderly with Chronic Diseases using Machine Learning Techniques.,” Healthc. Inform. Res., vol. 20, no. 2, pp. 125–34, 2014.
[13] J. Jonnagaddala, H. Dai, and P. Ray, “A preliminary study on automatic identification of patient smoking status in unstructured electronic health records,” no. BioNLP, pp. 147–151, 2015.
[14] C. M. Rochefort, A. D. Verma, T. Eguale, T. C. Lee, and D. L. Buckeridge, “A novel method of adverse event detection can accurately identify venous thromboembolisms (VTEs) from narrative electronic health record data.,” J. Am. Med. Inform. Assoc., vol. 22, no. 1, pp. 155–65, 2015.
[15] Y. Wu, J. Xu, Y. Zhang, and H. Xu, “Clinical Abbreviation Disambiguation Using Neural Word Embeddings,” no. BioNLP, pp. 171–176, 2015.
[16] T. Santhanam and M. S. Padmavathi, “Application of K-Means and Genetic Algorithms for Dimension Reduction by Integrating SVM for Diabetes Diagnosis,” Procedia Comput. Sci., vol. 47, pp. 76–83, 2015.
[17] L. Auria and R. A. Moro, “Support vector machines (SVM) as a technique for solvency analysis,” 2008.
[18] D. Meyer and F. H. T. Wien, “Support vector machines,” Interface to libsvm Packag. e1071, 2015.
[19] B. W. Matthews, “Comparison of the predicted and observed secondary structure of {T4} phage lysozyme,” Biochim. Biophys. Acta - Protein Struct., vol. 405, no. 2, pp. 442–451, 1975.