Modelo de minería de texto aplicado a historiales clínicos electrónicos de pacientes de cuidados paliativos en Panamá
Text mining model applied to electronic medical records of palliative care patients in Panama
Denis Cedeño-Moreno
Grupo de Investigación en Salud Electrónica y Supercomputación,
Universidad Tecnológica de Panamá
[email protected]
Miguel Vargas-Lombardo
Grupo de Investigación en Salud Electrónica y Supercomputación
Universidad Tecnológica de Panamá,
[email protected]
Resumen– La minería de texto se basa en la extracción de nuevo conocimiento a partir de datos no estructurados en lenguaje natural. La aplicación de técnicas de minería de texto para el dominio de la medicina, en especial de la información de los registros electrónicos de salud de los pacientes de cuidados paliativos, es una de las áreas más recientes y prometedores de investigación para el análisis de datos textuales. Además podemos crear ontologías para describir la terminología y el conocimiento en un dominio dado. En una ontología se formaliza la conceptualización de un dominio que puede ser general o específico. En el trabajo proponemos un modelo para encontrar patrones de información relevante en los registros electrónicos de salud de los pacientes de las unidades de cuidados paliativos en Panamá, basados en la utilización de las fases de la minería de texto y el desarrollo de una ontología para descubrir conocimiento oculto.
Palabras claves– Conocimiento, cuidados paliativos, historia clínica electrónica, minería de texto, ontología.
Abstract– Text mining is based on new knowledge extraction from unstructured natural language data. The application of techniques of text mining for the domain of medicine, especially information from electronic health records of patients in palliative care is one of the most recent and promising research areas for the analysis of textual data. We may also create ontologies to describe the terminology and knowledge in a given domain. In an ontology, conceptualization of a domain that may be general or specific formalized. In the paper, we propose a model to find patterns of relevant information in the electronic health records of patients in palliative care units in Panama, based on the use phase of text mining and development of an ontology to discover hidden knowledge.
Keywords– Knowledge, palliative care, electronic health record, text mining, ontology.
1.Introducción
Los seres humanos desde la antigüedad se han dedicado a recolectar en su momento semillas, frutas, herramientas, hoy nos hemos dado a la tarea de recolectar información. Dicha información la recolectamos en diferentes fuentes, ya sea en medios electrónicos o en papel común. Se sabe que el conocimiento [1], es un tesoro para los seres humanos y quien lo tiene posee el control de la situación.
Tener este conocimiento depende de la capacidad de los seres humanos de manejar ciertas tareas con la información, saber dónde buscarla, para resumir grandes volúmenes de información y hacer de este el conocimiento necesario [2].
Con el desarrollo de tecnologías relacionadas con la información se puede acceder y analizar cantidades de conocimiento relacionado con la salud. Muchas de estas herramientas se basan en la extracción de conocimiento a partir de fuentes de información textuales mediante la aplicación de la lingüística computacional. La lingüística computacional, como se indica [3], se centra principalmente en el diseño de mecanismos que permiten a los computadores entender el lenguaje natural, así como diversas tareas de procesamiento de la información.
La minería de texto (MT) es una aplicación de la lingüística computacional y procesamiento de textos que tiene por objeto facilitar la identificación y extracción de nuevos conocimientos a partir de colecciones de documentos o corpus de textos.
El objetivo principal de este trabajo es presentar el diseño de un modelo para la extracción de conocimiento, basado en los procesos de la MT y el desarrollo de una ontología, y que a través de esta nos permita encontrar patrones y obtener información de las historias clínicas de los pacientes de unidades de cuidados paliativos en la República de Panamá. El resto de este manuscrito es estructurado de la siguiente manera: En la siguiente sección se describe la metodología para hacer este trabajo. Luego se muestran las experiencias del diseño del modelo propuesto. Después de eso, se presenta una discusión. Por último, señalamos las principales conclusiones de este trabajo.
2. Materiales y Métodos
2.1 Minería de Texto
Cada día una enorme cantidad de datos son generados [4,5] en las organizaciones de salud [6], es necesario el diseño y desarrollo de nuevas y potentes herramientas de procesamiento de la información, con el avance de las tecnologías relacionadas con la información se pueden acceder y analizar estos datos todos ellos relacionados con los registros electrónicos de salud del paciente [7].
Por lo tanto, es común hoy día para un especialista registrar los datos del paciente por vía electrónica [8], esto incluye no solo la información general del paciente, sino también lo relacionado con el diagnóstico, los resultados analíticos, pruebas funcionales y la medicación [9]. Manteniendo esta gran cantidad de información en formato digital se tienen tres ventajas principales: se mejora la calidad de la atención, se reduce el tiempo de trabajo del personal de salud, y se puede utilizar dicha información a través de sistemas automatizados, tales como la minería de textos [10].
MT [11,12] es el área de investigación más reciente del procesamiento de textos. Se define como el proceso de descubrir patrones interesantes y nuevos conocimientos en una colección de textos. Es decir la MT es responsable de descubrir nuevo conocimiento que no existía explícitamente en los textos [13]. Solo los computadores pueden manipular rápidamente grandes cantidades de texto.
El proceso de MT consta de dos fases principales: pre procesamiento y la fase de descubrimiento [14]. En la primera fase, los textos se convierten en una especie de representación estructurada o semiestructurada a continuación se facilita el análisis, mientras que en la segunda fase las representaciones intermedias son analizadas con el fin de descubrir en ellas algunos patrones interesantes o nuevo conocimiento.
La MT es un área multidisciplinar [15], que ha experimentado un aumento exponencial en la producción de información. Junto con la tecnología de la información han dado lugar a la gestión de sistemas complejos y suministros de información para diversas tareas. Debido a que la mayor parte de la información (más del 80%) se encuentra actualmente almacenada en forma de texto, se cree que la MT [16], tiene un gran valor comercial y organizacional.
Otro contexto de gran importancia es la utilización de las herramientas de MT para la formación y la investigación. En estas áreas, hay una enorme cantidad de fuentes de información, difícil de manejar y seleccionar. De hecho, hay muchas publicaciones en papel que tratan de reunir lo más relevante que se produce en la publicación en determinadas áreas del conocimiento médico.
Procesar grandes volúmenes de texto no estructurado para extraer el conocimiento requiere la aplicación de una serie de técnicas que incluyen la Recuperación de Información (RI), Procesamiento del Lenguaje Natural (PLN) [17,18,19,20], y la Extracción de Información (EI).
Los sistemas de RI identifican los documentos de una colección que coincide con la consulta del usuario, son los motores de búsqueda más populares utilizados por ejemplo en Google, que identifican los documentos en la World Wide Web que son relevantes para un determinado conjunto de palabras.
PLN es uno de los temas más antiguos y más difíciles en el campo de la inteligencia artificial. Es el análisis del lenguaje humano para lograr que las computadoras entiendan el lenguaje natural como lo hacen los humanos, el papel del PLN en la MT es proporcionar sistemas para la extracción de información de forma tal que los datos lingüísticos que se necesitan para realizar su tarea sean lo más adecuados posible.
EI es el proceso de obtención automática de un documento estructurado a partir de datos del lenguaje natural. A menudo se trata de definir la forma general de la información que nos interesa como una o más plantillas, que luego se utilizan para guiar el proceso de extracción.
La información médica que está registrada en la Historia Clínica Electrónica (HCE) del paciente es muy valiosa. Además de su uso como parte de la historia del paciente en las unidades de cuidados paliativos, las HCE, pueden ser tratadas como un repositorio de información del paciente y proporcionarnos datos ricos. Las HCE están solo a la espera de ser analizadas y procesadas para el descubrimiento del conocimiento clínico.
2.2 Minería de Texto e Historia Clínica Electrónica
Existe una importante fuente de datos que se puede utilizar para la investigación y para mejorar la calidad de los servicios de salud, inmersas en las HCE [21,22]. Las HCE han estado disponibles y debido a su nivel de detalle, están ganando aceptación para el uso de herramientas tecnológicas de procesamiento de la información.
Los médicos escriben sus comentarios en las HCE y varían de un sistema a otro, sin embargo, se suelen almacenar los siguientes datos: edad, sexo, diagnóstico, historial médico, los medicamentos con receta, exámenes de laboratorio, procedimientos clínicos, resultados, alergias, inmunizaciones, signos y observaciones vitales [23]. La información contenida puede estar en forma narrativa o forma semiestructurada [24,25]. Con ellos, se pueden realizar análisis y extraer información con el fin de mejorar tanto las tareas de la investigación médica y científica, así como los procedimientos al paciente.
Además de su uso clínico por el médico, las HCE, se puede utilizar como un repositorio para la información médica [26]. Los investigadores médicos [27] están en el umbral de una nueva era en la que las HCE están ganando un papel importante en el apoyo a las actividades diarias. Las herramientas de la informática y la gestión del conocimiento [28], son ahora parte del mundo de la ciencia biomédica [29].
Plataformas e infraestructuras de computación permiten nuevos tipos de experimentos que eran imposibles de hacer hace diez años. Los avances en el área de tecnología de información para la salud (HIT) [30], sin duda, han permitido avances en la documentación del paciente. Los avances en HIT sin duda han transformado la forma en que se lleva a cabo el cuidado de la salud, sino que también ha cambiado la forma en que se está documentando datos de los pacientes [31]. Esta generación de los datos de salud electrónica es una gran promesa para contribuir significativamente a la salud de los pacientes, y también para transformar la investigación biomédica [32].
Hay un progreso significativo en los últimos años en la aplicación de técnicas de la MT y las HCE con el fin de hacer frente al gran volumen de información en el área de rápido crecimiento de la literatura médica. Sin embargo, es raro encontrar aplicaciones de MT en el área de sistemas de información clínica, el principal desafío para la MT en aplicaciones médicas en los próximos años es hacer este tipo de sistemas útiles para los investigadores. La MT ha surgido como una solución potencial para las HCE de pacientes que sufren de enfermedades tales como sistemas de cáncer [33].
2.3 Minería de Texto y HCE de pacientes con cáncer
La MT puede ayudar a adquirir conocimientos de una montaña de texto y su uso está hoy ampliamente extendido en la investigación biomédica [34]. Muchos investigadores han utilizado la tecnología MT para descubrir nuevos conocimientos y mejorar el desarrollo de la investigación biomédica, especialmente las relacionadas con enfermedades malignas como el cáncer.
Hay un recurso valioso en el contexto biomédico que podemos utilizar y se permite con mucho texto actual de los pacientes con cáncer que reciben tratamiento en hospitales en el país y que se almacenan en la HCE.
El cáncer es una enfermedad mortal que causó 7,4 millones de muertes en 2008 [35]. Por esta razón, el cáncer es una de las áreas más importantes de estudio para los investigadores biomédicos. Con tanto texto sobre esta enfermedad, es casi imposible para los médicos investigar todos estos documentos y descubrir nuevos conocimientos que sean significativos. La MT puede ayudar a los investigadores para completar esta difícil tarea.
El investigador puede constatar las ventajas de la MT y facilitar la investigación para ayudar en la búsqueda de nuevos conocimientos, para el diagnóstico, tratamiento y prevención del cáncer [36].
La MT emplea muchas de las tecnologías informáticas, tales como el aprendizaje automático, inteligencia artificial, bioestadística, tecnología de la información, y el reconocimiento de patrones para encontrar nuevos resultados ocultos en texto de biomédica [37].
En los últimos años, la MT y el análisis estadístico [38] se han aplicado a grandes áreas de la medicina, debido a la existencia de grandes volúmenes de datos. El uso de estas herramientas permiten que el trabajo de los médicos y las decisiones sean más fáciles y más precisas, mejorando así el servicio que ofrecen, también proporcionan un mecanismo para transformar el texto en conocimiento. Por tal razón hemos decidido aplicar en nuestro proyecto algunas técnicas de MT para explorar, analizar, consultar y gestionar los datos de pacientes con cáncer del área de cuidados paliativos [39].
3. Escenario de estudio
3.1 Representación del conocimiento
Diversas áreas de convergencia del conocimiento [40], han llevado al diseño e implementación un sinnúmero de sistemas informáticos que apoyan la integración de bases de datos con información médica.
La informática médica tiene décadas de experiencia en el desarrollo de aplicaciones y en el procesamiento de la información de pacientes, y ha permitido contribuir al desarrollo de herramientas innovadoras en el campo tecnológico e incluye distintas áreas como la recuperación y la lingüística computacional [41]. A la extracción de conocimiento con el lenguaje natural y almacenamiento en bases de datos textuales, se conoce comúnmente como descubrimiento de conocimiento en texto (KDT) [42].
3.2 Rol del especialista en cuidados paliativos
Actualmente los médicos especialistas de cuidados paliativos tienen un registro completo de las actividades e interacciones de los pacientes y otros médicos involucrados en el proceso de evaluación [43]. Un análisis más detallado de este conjunto de actividades e interacciones nos permite comprender lo que ocurrió con el paciente. Sin embargo, cuando se tiene una cantidad considerable de interacciones el análisis manual es prácticamente imposible debido a la tarea, el tiempo y el esfuerzo.
La aplicación de técnicas MT [44], al dominio de cáncer es una de las zonas más nuevas y más prometedoras de la investigación para el análisis de los datos [45].
3.3 Modelo propuesto
Nuestra herramienta permite analizar los elementos de texto de las HCE de pacientes de cuidados paliativos con el fin de identificar y ampliar los conocimientos de los especialistas.
Las funciones que debe cumplir principalmente una herramienta de MT incluyen: Identificar “hechos” y datos puntuales a partir del texto de los documentos; agrupar documentos similares lo que también se conoce como clustering; determinar temas abordados en los documentos mediante la categorización automática de los textos; identificar los conceptos tratados en los documentos y crear redes de conceptos; facilitar el acceso a la información repartida entre los documentos de la colección; visualización y navegación de colecciones de texto.
En nuestro proyecto de descubrir el conocimiento de los documentos de texto en los HCE de los pacientes de cuidados paliativos, debemos pasar por varias etapas importantes que inducirá este proceso [46]. Estas etapas se pueden ver en la figura 1.
Pre-Proceso: n este paso se seleccionan aquellos términos que mejor representan los objetos de estudio, también se elimina información irrelevante y se llevarán a cabo las operaciones o transformaciones en el texto, para generar una representación semi estructurada lo que debe contribuir a facilitar el análisis.
Descubrimiento: sta es la etapa en la que se analizarán las representaciones intermedias con el fin de descubrir en ellos algunos patrones interesantes o nuevos conocimientos, aplicando técnicas y algoritmos de minería.
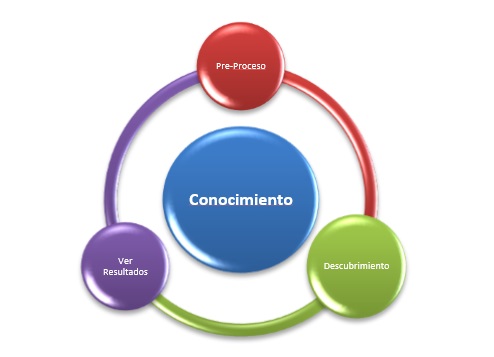
Figura1. Etapas de la Minería de Texto.
Una de las etapas más importantes y críticas de la MT es la estructuración del contenido, es decir lograr una representación o modelo intermedio del texto [47] sobre esta base es que pueden aplicarse los algoritmos o métodos de descubrimiento como las ontologías.
La forma más comúnmente utilizada hoy en día para la estructuración de contenido es el modelo de espacio vectorial, así como taxonomías de conceptos y grafos [48].
Consideramos hacer frente en este proyecto al proceso de la estructuración del contenido manejándolo con la aplicación de un nuevo modelo, ya que encontrar nuevas alternativas para la representación o estructuración de textos [49], permite la innovación en la recuperación de la información, y logra que se faciliten los procesos que nos lleven a descubrir el conocimiento.
El proceso de la estructuración [50], lo manejaremos en varias fases, la primera fase del nuevo modelo, estará basada en una aplicación desarrollada en lenguaje de programación Java, con la utilización de clases especializadas para separar o romper los elementos de una cadena de texto, la tokenización, que recibe como entrada el archivo de texto y produce una salida compuesta de tokens o símbolos.
En este sentido, se define una primera aproximación de un modelo de representación de textos, así como un método para su construcción de forma automática. Este modelo se puede ver en la figura 2.
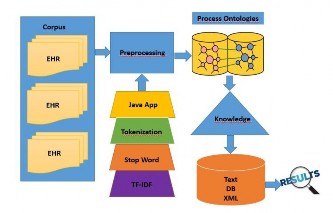
Figura 2. Modelo propuesto.
En el modelo la entrada está dada por un archivo de texto con el corpus de lo que se desea procesar, este archivo de texto contendrá los comentarios hechos por los médicos especialistas de cuidados paliativos, específicamente el área donde se especifican las observaciones realizadas al paciente en la HCE.
Como se ha señalado dentro de la HCE de cada paciente que asiste a la unidad de cuidados paliativos y es atendido por un médico especialista, se detallan cada una de estas observaciones. Deseamos reunir una muestra considerable de un grupo de pacientes para ser almacenadas en este archivo (corpus) que funcionará como elemento de entrada al proceso.
Una vez obtenido el archivo y a través de una aplicación hecha en lenguaje de programación Java y utilizando herramientas como las clases de tokenización leeremos el archivo al cual lo iremos separando en unidades básica (palabra a palabra) hasta formar una nueva estructura.
Esta estructura nueva que contendrá los diferentes elementos de nuestro corpus la almacenaremos en un archivo de texto. Luego pasará por una fase que logre eliminar los conceptos o elementos con menor importancia o relevancia para nosotros, debido a que no todas las palabras de nuestras HCE son igualmente representativas del corpus que vamos a evaluar.
Lo que en esencia estamos realizando es un análisis léxico de nuestro texto inicial, cuyo propósito es el tratamiento de números, guiones, signos de puntuación, palabras mayúsculas o minúsculas, nombres propios. Luego son eliminados los conceptos que tengan menor relevancia, en cuanto a su vínculo contextual con el resto de los conceptos, es lo que se conoce como palabras vacías.
Lo que tenemos es una lista de palabras relevantes para nuestro proyecto, a cada palabra le vamos a asignar distintos pesos. Para ello vamos a utilizar las técnicas del modelo de ponderación TF-IDF (term frequency– inverse document frequency) [51] para poder evaluar la importancia de cada palabra en el corpus de documentos.
En la segunda fase nos vamos a enfocar en deducir el conocimiento a través de la identificación de patrones e información. Revelando conocimiento implícito que no es perceptible desde un solo documento, sino a partir de una serie de documentos.
Es una realidad que hoy una gran cantidad de herramientas de MT utilizan las ontologías [52] para la búsqueda de conocimiento. En las ontologías, las palabras describen conceptos que definen de manera formal las relaciones y las reglas que especifican las dependencias entre los conceptos. Las ontologías se utilizan para estructurar y categorizar la información específica de un dominio.
Muchas aplicaciones TM han implementado ontologías [53] en sus flujos de trabajo para estructurar su estrategia de búsqueda, visualización y clasificación de la información.
Consideramos que desarrollando una ontología específica con la información de los HCE de pacientes de cuidados paliativos lograremos la recuperación de conocimientos [54] de este corpus. Las ontologías permiten la expansión de consultas, la reformulación de una consulta para mejorar el rendimiento de la recuperación de palabras clave.
La utilización de una ontología [55] en el proyecto, se enfocará en organizar de manera sistemática el conocimiento a partir de un conjunto de términos, conceptos y relaciones entre ellos. Debido a que las ontologías definen y establecen relaciones complejas, incorporan reglas y axiomas que no tienen otros elementos de naturaliza lingüísticos, obteniendo una representación formal de los conceptos y las relaciones existentes entre ellos.
4. Conclusión y trabajos futuros
Se dispone en la actualidad de gran cantidad de información en el área médica como bien se ha señalado. Sin embargo, existe un problema inherente a tal volumen de datos: su procesamiento. Ya sea a través de la recuperación selectiva o la interpretación se hacen prácticamente imposibles para un profesional si emplea los métodos clásicos.
Lo que hace vital la construcción de herramientas tecnológicas que lo ayuden en estas tareas. En ese escenario, el uso de herramientas como la minería de textos o las ontologías adquiere una relevancia fundamental. Estas herramientas, que han tenido ya un papel importante en otros campos del saber, se han empezado a utilizar recientemente en la medicina.
Como trabajo futuro se tiene contemplado el análisis de los algoritmos de MT, así como el desarrollo de la ontología que logre la extracción de conocimiento.
5. Reconocimiento
Nuestro agradecimiento al apoyo del Grupo de Electrónica de Investigación y Salud de Supercomputación de la Universidad Tecnológica de Panamá, especialmente su director el Dr. Miguel Vargas-Lombardo por su asesoramiento.
6. Referencias
[1] S. M. Allameh and S. M. Zare, “Examining the impact of KM enablers on knowledge management processes”, Procedia Comput. Sci, vol. 3, pp. 1211–1223, 2011.
[2] M. Terzieva, “Project Knowledge Management: How Organizations Learn from Experience”, Procedia Technol, vol. 16, pp. 1086–1095, 2014.
[3] K. a. Dill, A. Lucas, J. Hockenmaier, L. Huang, D. Chiang, and A. K. Joshi, “Computational linguistics: A new tool for exploring biopolymer structures and statistical mechanics” Polymer (Guildf), vol. 48, no. 15, pp. 4289–4300, 2007.
[4] N. Zhong, S. Member, Y. Li, and S. Wu, “Effective pattern discovery for text mining”, 2010.
[5] H. Hashimi, A. Hafez, and H. Mathkour, “Selection criteria for text mining approaches”, Comput. Human Behav, vol. 51, pp. 729– 733, 2015.
[6] T. Suzuki, H. Yokoi, S. Fujita, and K. Takabayashi, “Automatic DPC code selection from electronic medical records: Text mining trial of discharge summary”, Methods Inf. Med, vol. 47, pp. 541– 548, 2008.
[7] Y. H. Tseng, C. J. Lin, and Y. I. Lin, “Text mining techniques for patent analysis” Information Processing & Management. vol. 43, no. September, pp. 1216–1247, 2007.
[8] S. Ananiadou, D. B. Kell, and J. Tsujii, “Text mining and its potential applications in systems biology.” Trends Biotechnol. vol. 24, no. 12, pp. 571–579, 2006.
[9] S. Ananiadou and J. McNaught, “Text Mining for Biology and Biomedicine” Comput. Linguist. vol. 33, pp. 135–140, 2006.
[10] G. Akçapinar, “How automated feedback through text mining changes plagiaristic behavior in online assignments” Comput. Educ., vol. 87, pp. 123–130, 2015.
[11] Q. Mei and C. Zhai, “Discovering evolutionary theme patterns from text: an exploration of temporal text mining” Proc. Elev. ACM SIGKDD Int. Conf. Knowl. Discov. data Min, pp. 198–207, 2005.
[12] H.-C. Yang, C.-H. Lee, and H.-W. Hsiao, “Incorporating SelfOrganizing Map with Text Mining Techniques for Text Hierarchy Generation” Appl. Soft Comput, vol. 34, pp. 251–259, 2015.
[13] J. I. Guerrero, C. León, I. Monedero, F. Biscarri, and J. Biscarri, “Improving Knowledge-Based Systems with statistical techniques, text mining, and neural networks for non-technical loss detection” Knowledge-Based Syst, vol. 71, pp. 376–388, 2014.
[14] H. Mahgoub, H. Mahgoub, N. Ismail, N. Ismail, F. Torkey, and F. Torkey, “A Text Mining Technique Using Association Rules Extraction” World Health, pp. 21–28, 2008.
[15] H. (National U. of S. Liu, H. (Osaka U. Motoda, R. Setiono, and Z. Zhao, “Feature Selection an ever evolving frontier in Data Mining” J. Mach. Learn. Res. Work. Conf. Proc. 10 Fourth Work. Featur. Sel. Data Min., pp. 4–13, 2010.
[16] M. Reinberger and P. Spyns, “Unsupervised text mining for the learning of dogma-inspired ontologies” Ontol. Learn. from Text Methods, Appl. Eval, no. September, pp. 29–43, 2005.
[17] M. Krallinger, R. a a Erhardt, and A. Valencia, “Text-mining approaches in molecular biology and biomedicine” Drug Discov. Today, vol. 10, no. 6, pp. 439–445, 2005.
[18] T. Baldwin, P. Cook, B. Han, A. Harwood, S. Karunasekera, and M. Moshtaghi, A Support Platform for Event Detection using Social Intelligence. 2012.
[19] Y. Yang, L. Akers, T. Klose, and C. Barcelon Yang, “Text mining and visualization tools - Impressions of emerging capabilities” World Pat. Inf., vol. 30, no. September 2015, pp. 280– 293, 2008.
[20] A. Stavrianou, P. Andritsos, and N. Nicoloyannis, “Overview and semantic issues of text mining” ACM SIGMOD Rec., vol. 36, no. 3, p. 23, 2007.
[21] D. J. Berndt, J. a. McCart, D. K. Finch, and S. L. Luther, “A Case Study of Data Quality in Text Mining Clinical Progress Notes” ACM Trans. Manag. Inf. Syst., vol. 6, no. FEBRUARY, pp. 1–21, 2015.
[22] P. Lependu, S. V Iyer, C. Fairon, and N. H. Shah, “Annotation Analysis for Testing Drug Safety Signals using Unstructured Clinical Notes”, J. Biomed. Semantics, vol. 3 Suppl 1, no. Suppl 1, p. S5, 2012. [23] S. L. West, W. Johnson, W. Visscher, M. Kluckman, Y. Qin, and A. Larsen, “The challenges of linking health insurer claims with electronic medical records”, Health Informatics J., vol. 20, pp. 22– 34, 2014.
[24] R. Cohen, M. Elhadad, and N. Elhadad, “Redundancy in electronic health record corpora: analysis, impact on text mining performance and mitigation strategies”, BMC Bioinformatics, vol. 14, p. 10, 2013.
[25] S. Tanuja, D. Acharya, and K. R. Shailesh, “Comparison of different data mining techniques to predict hospital length of stay” J. Pharm. Biomed. Sci., vol. 07, no. 07, 2011. [26] N. Ramakrishnan, D. Hanauer, B. Keller, and B. Ramakrishnan, N., Hanauer, D., & Keller, “Mining electronic health records” Computer (Long. Beach. Calif). vol. 43, no. October, pp. 77–81, 2010.
[27] M. Kvist, M. Skeppstedt, S. Velupillai, and H. Dalianis, “Modeling human comprehension of Swedish medical records for intelligent access and summarization systems - Future vision, a physician perspective” 9th Scand. Conf. Heal. Informatics, 2011.
[28] I. Fatima, M. Fahim, D. Guan, Y.-K. Lee, and S. Lee, “Socially interactive CDSS for u-life care” Proc. 5th Int. Confernece ubiquitous Inf. Manag. Commun., no. September 2015, pp. 1–8, 2011.
[29]R. Harpaz, A. Callahan, S. Tamang, Y. Low, D. Odgers, S. Finlayson, K. Jung, P.LePendu, and N. H. Shah, “Text Mining for Adverse Drug Events: the Promise, Challenges, and State of the Art” Drug Saf., no. September 2015, 2014.
[30] Holzinger, R. Geierhofer, F. Mödritscher, and R. Tatzl, “Semantic Information in Medical Information Systems: Utilization of Text Mining Techniques to Analyze Medical Diagnoses” J. Univers. Comput. Sci., vol. 14, no. 22, pp. 3781–3795, 2008.
[31] M. Andrade-Navarro and C. Perez-Iratxeta, “Text mining of biomedical literature: Doing well, but we could be doing better” Methods, vol. 74, pp. 1–2, 2015.
[32] R. a A. Seoud and M. S. Mabrouk, “TMT-HCC: A tool for text mining the biomedical literature for hepatocellular carcinoma (HCC) biomarkers identification” Comput. Methods Programs Biomed. vol. 112, no. 3, pp. 640–648, 2013.
[33] I. Spasic, J. Livsey, J. a Keane, and G. Nenadic, “Text mining of cancer-related information: Review of current status and future directions” Int. J. Med. Inform., vol. 83, pp. 605–623, 2014.
[34] B. Xie, Q. Ding, H. Han, and D. Wu, “MiRCancer: A microRNA-cancer association database constructed by text mining on literature” Bioinformatics, vol. 29, pp. 638–644, 2013.
[35] Y.-C. Fang, H.-C. Huang, and H.-F. Juan, “MeInfoText: associated genemethylation and cancer information from text mining” BMC Bioinformatics, vol. 9, p. 22, 2008.
[36] Who, “World Health Statistics 2009” p. 149, 2009.
[37] A. Korhonen, I. Silins, L. Sun, and U. Stenius, “The first step in the development of Text Mining technology for Cancer Risk Assessment: identifying and organizing scientific evidence in risk assessment literature.” BMC Bioinformatics, vol. 10, p. 303, 2009.
[38] R. Jelier, M. J. Schuemie, A. Veldhoven, L. C. J. Dorssers, G. Jenster, and J. a Kors, “Anni 2.0: a multipurpose text-mining tool for the life sciences” Genome Biol., vol. 9, no. 6, p. R96, 2008.
[39] H. C. Beck, Mass spectrometry in epigenetic research, vol. 593. 2010.
[40] H. Chen, R. H. L. Chiang, and V. C. Storey, “Business Intelligence and Analytics: From Big Data to Big Impact” Mis Q., vol. 36, no. 4, pp. 1165–1188, 2012.
[41] D. Chiang, “Hierarchical Phrase-Based Translation” no. May 2006, 2007.
[42] W. W. M. Fleuren and W. Alkema, “Application of text mining in the biomedical domain” Methods, vol. 74, pp. 97–106, 2015.
[43] N. B. Ngwenya and S. Mills, “The use of weblogs within palliative care: A systematic literature review” Health Informatics J., vol. 20, pp. 13–21, 2014. [44] W. Der Yu and J. Y. Hsu, “Content-based text mining technique for retrieval of CAD documents” Autom. Constr., vol. 31, pp. 65–74, 2013.
[45] A. Jimeno Yepes and R. Berlanga, “Knowledge based wordconcept model estimation and refinement for biomedical text mining” J. Biomed. Inform. vol. 53, pp. 300–307, 2014.
[46] A. Hotho, A. Nürnberger, and G. Paaß, “A Brief Survey of Text Mining” LDV Forum - Gld. J. Comput. Linguist. Lang. Technol, vol. 20, pp. 19–62, 2005. [47] V. C. Pande and A. S. Khandelwal, “A Survey Of Different Text Mining Techniques,” IBMRD’s J. Manag. Res., vol. 3, no. 1, pp. 125–133, 2014.